转自武汉大学新闻网 网讯(通讯员杨雪)10月17日,国际期刊Nature Communications(《自然•通讯》)在线发表生命科学学院和高等研究院朱玉贤院士、周宇教授课题组在解析亚洲棉转录组方面联合取得的最新研究成果。该工作是迄今为止科学家对亚洲棉基因组的复杂转录所进行的最为准确的注释。
论文题为“Multi-strategic RNA-seq analysis reveals a high-resolution transcriptional landscape in cotton”(《应用多RNA组学策略精细解析亚洲棉基因组转录全景》)。生命科学学院副研究员王坤、周宇实验室博士研究生王得和为共同第一作者,朱玉贤、周宇为共同通讯作者,武汉大学为第一完成单位。该工作得到了国家自然科学基金、国家科技部、武汉大学自主科研交叉学科项目等的资助。
棉花是一种重要的天然纤维作物,也是研究细胞分化、伸长和细胞壁发育调控的重要模式植物,但缺乏全面、高分辨率的基因图谱,棉花转录组的不完整性阻碍了各种生物过程分子机制的研究。亚洲棉(G. aboreum)是现在全世界广泛种植的栽培棉种-陆地棉祖先基因组供体种之一,其基因组由1.7G碱基对组成。课题组通过系统地、全方位地转录分析阐释亚洲棉基因组,填补了国际上棉花基因组精细转录注释的空白,获得了迄今为止科学家对亚洲棉基因组的复杂转录全貌所进行的最准确的注释,为进一步的功能基因组研究提供了宝贵的资源。
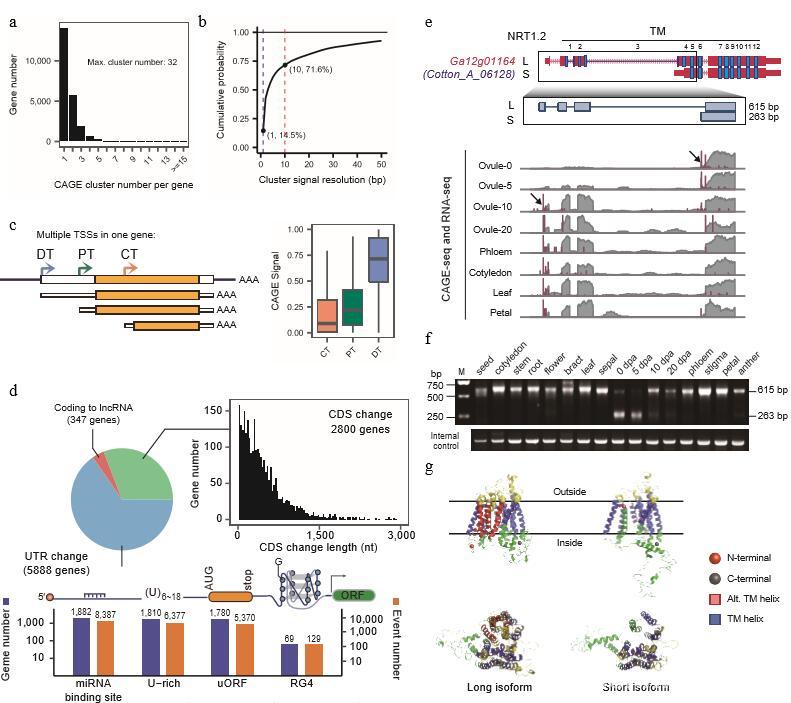
在研究中,团队引入并整合运用了四种高通量技术,包括Pacbio long reads Iso-seq、strand-specific RNA-seq、CAGE-seq和PolyA-seq,系统地探索了亚洲棉的16种组织或不同器官类型的精细转录组;首创开发运用了集成注释工具IGIA,从整合的数据中重建棉花基因组精确的基因结构和转录本异构体。
课题组在研究中还发现棉花基因组中存在高度动态的转录调控,如可变启动子和终止子调节、微外显子剪接、多顺反子转录通读和RNA选择性剪接热区等复杂现象。课题组进一步验证和探究了在亚洲棉基因组中所发现的这一类新颖的基因调控现象的分子机制和可能的生物学功能。此外,通过与已经发表的有关棉花重要农艺性状的遗传位点(GWAS和其他SNP)的比对分析发现,大量位点位于基因的非翻译区、RNA加工元件等区域。因此该项研究取得的重要发现对未来棉花功能基因组学的进一步发展意义重大。
该研究是朱玉贤课题组继雷蒙德氏棉基因组(Wang et al., 2012,Nature Genetics),亚洲棉基因组(Li et al., 2014,Nature Genetics;Du et al., 2018, Nature Genetics)和陆地棉基因组(Li et al. 2015, Nature Biotechnology)的组装和解析之后在棉花组学研究方面的又一重要突破。



 武大主页
武大主页 武大邮箱
武大邮箱 信息门户
信息门户 English
English